Cartografía genética
|
Read other articles:

Raionul Ialoveni adalah distrik di Moldova. Pusat administrasinya berada di Ialoveni. Pada 1 Januari 2005 jumlah penduduknya 97.500 orang. Artikel bertopik Eropa ini adalah sebuah rintisan. Anda dapat membantu Wikipedia dengan mengembangkannya.lbs

Vasa MuseumVasamuseetExterior of the Vasa Museum.Didirikan1990LokasiGalärvarvsvägen 14, Djurgår, SwedenJenismaritime museumWisatawan1,143,404 (2008)[1]DirekturMarika HedinSitus webwww.vasamuseet.se Museum Vasa (Swedia: Vasamuseetcode: sv is deprecated ) merupakan museum maritim yang terletak di Stockholm, Swedia. Letaknya di Pulau Djurgåden. Museum ini dibuka tahun 1990 dan kunjungan terbesar museum di Skandinavia. Catatan ^ Årsredovisning 2008. Statens maritima museer. Diarsipka...

Biografi ini memerlukan lebih banyak catatan kaki untuk pemastian. Bantulah untuk menambahkan referensi atau sumber tepercaya. Materi kontroversial atau trivial yang sumbernya tidak memadai atau tidak bisa dipercaya harus segera dihapus, khususnya jika berpotensi memfitnah.Cari sumber: Abdul Hamid II – berita · surat kabar · buku · cendekiawan · JSTOR (Pelajari cara dan kapan saatnya untuk menghapus pesan templat ini) Untuk kegunaan lain, lihat Abdul H...

2012 2022 Élections législatives françaises de 2017 577 députés de l'Assemblée nationale(majorité absolue : 289 sièges) 11 et 18 juin 2017 Corps électoral et résultats Votants au 1er tour 23 167 508 48,70 % 8,5 Votants au 2d tour 20 164 615 42,64 % 12,8 Majorité présidentielle – Édouard Philippe Liste La République en marcheMouvement démocrateMouvement des progressistesParti écologiste Voix au 1er to...

Artikel ini tidak memiliki referensi atau sumber tepercaya sehingga isinya tidak bisa dipastikan. Tolong bantu perbaiki artikel ini dengan menambahkan referensi yang layak. Tulisan tanpa sumber dapat dipertanyakan dan dihapus sewaktu-waktu.Cari sumber: Pemangku kepentingan – berita · surat kabar · buku · cendekiawan · JSTOR Artikel atau sebagian dari artikel ini mungkin diterjemahkan dari Stakeholder (corporate) di en.wikipedia.org. Isinya masih belum ...

Aspect of the South-East Asian Theatre of World War II For action after November 1944, see Burma campaign (1944–1945). Burma Campaign 1943–1944Part of the Pacific War during World War IIA C-47 transport aircraft drops supplies by parachute to Allied troops in action against Japanese forces; a common event during the fighting in Burma and India during 1944.DateOctober 1943 – November 1944LocationState of BurmaAssam, British IndiaYunnan, Republic of ChinaResult Allied victoryBelligerents ...

Halaman ini berisi artikel tentang istilah ekonom. Untuk album kompilasi karya musisi Inggris Aim, lihat Means of Production. Artikel ini bukan mengenai Mode produksi. Ekonomi Ekonomi menurut kawasan Afrika · Amerika Amerika Selatan · Asia Eropa · Oseania Kategori umum Ekonomi mikro · Ekonomi makro Sejarah pemikiran ekonomi Metodologi · Pendekatan heterodoks Bidang dan subbidang Perilaku · Budaya · ...

فرانسوا إدوار بيكو (بالفرنسية: François-Édouard Picot) معلومات شخصية الميلاد 17 أكتوبر 1786(1786-10-17)باريس الوفاة 15 مارس 1868 (81 سنة)باريس مكان الدفن مقبرة بير لاشيز[1] مواطنة فرنسا[2] عضو في أكاديمية الفنون الجميلة[1] الحياة العملية المدرسة الأم المدرسة الوطنية للف...

Coppa dell'Unione Sovietica 1951Kubok SSSR 1951 Competizione Kubok SSSR Sport Calcio Edizione 12ª Organizzatore FFSSSR Date dal 12 agosto 1951al 17 ottobre 1951 Luogo Unione Sovietica Partecipanti 51 Formula turni ad eliminazione diretta Risultati Vincitore CDSA Mosca(3º titolo) Secondo Città di Kalinin Semi-finalisti VVS Mosca Šachtyor Stalino Statistiche Incontri disputati 52 Gol segnati 202 (3,88 per incontro) Cronologia della competizione 1950 1952...

Paul ReubensReubens pada 2019LahirPaul Rubenfeld(1952-08-27)27 Agustus 1952Peekskill, New York, A.S.Meninggal30 Juli 2023(2023-07-30) (umur 70)Los Angeles, California, A.S.PendidikanUniversitas BostonInstitut Seni California (BFA)PekerjaanAktorkomedianpenulisproduserTahun aktif1977–2023Orang tuaMilton Rubenfeld (ayah)KerabatAbby Rubenfeld (adik)Situs webpeewee.com Paul Reubens (/ˈruːbənz/; lahir Rubenfeld; 27 Agustus 1952 – 30 Juli 2023) adalah seorang aktor da...

Japanese engineer Ryoichi Nakagawa中川 良一Ryoichi Nakagawa at the age of 26 in 1939 or 1940Born(1913-04-27)27 April 1913Tokyo, JapanDied30 July 1998(1998-07-30) (aged 85)NationalityJapaneseEducationTokyo Imperial UniversityOccupationEngineerEngineering careerDisciplineMechanical engineeringSignificant designNakajima Homare engine (Ha-45)Significant advanceNakajima Sakae engineAwardsJapan Automotive Hall of Fame (2018) [1] Ryoichi Nakagawa (中川 良一, Nakagawa Ryōichi, 1...

The Misconceptions of UsAlbum kompilasi karya SHINeeDirilis8 Agustus 2013Direkam2012–2013 di S.M. Studios, Seoul, Korea Selatan.GenreK-pop,[1] Electropop,[2] Dance[3] World music[4]Durasi34:56 (disc satu)37:36 (disc dua)BahasaKoreaLabelS.M. Entertainment (record)KT Music (distribusi)ProduserLee Soo Man (executive)Kronologi SHINee Boys Meet U(2013)Boys Meet U2013 The Misconceptions of Us(2013) Everybody(2013)Everybody2013 The Misconceptions of Us adalah al...

Telê Santana Telê Santana nel 1993 Nazionalità Brasile Altezza 176 cm Peso 61 kg Calcio Ruolo Allenatore (ex ala destra) Termine carriera 1963 - giocatore1996 - allenatore CarrieraSquadre di club1 1951-1960 Fluminense557 (162)1960-1962 Guarani? (?)1963 Vasco da Gama? (?)Carriera da allenatore 1969-1970 Fluminense1971 Atlético Mineiro1972 San Paolo1973-1976 Atlético Mineiro1977-1979 Grêmio1980 Palmeiras1980-1982 Brasile1983-1985 Al...

Indonesian statesman and economist (1911–1989) Sjafruddin PrawiranegaraSjafruddin in 1947Prime Minister of the Revolutionary Government of the Republic of IndonesiaIn office15 February 1958 – 25 August 1961Preceded byOffice establishedSucceeded byOffice abolishedChairman of the Emergency Government of the Republic of Indonesia[a]In office22 December 1948 – 13 July 1949Preceded bySukarnoSucceeded bySukarno[b]3rd Deputy Prime Minister of IndonesiaIn o...
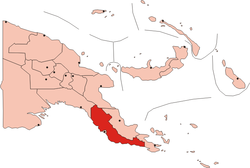
Artikel ini bukan mengenai Provinsi Papua Tengah. Koordinat: 9°30′S 147°40′E / 9.500°S 147.667°E / -9.500; 147.667 Provinsi Tengah Central ProvinceProvinsi BenderaCountry Papua New GuineaIbukotaBautamaPemerintahan • GubernurRufina PeterLuas • Total11,582 sq mi (29.998 km2)Populasi (2011) • Total269.756 • Kepadatan23/sq mi (9,0/km2)Zona waktuUTC+10 (AEST) Provinsi Tengah adalah sebuah ...

Pernikahan adalah salah satu jenis upacara Upacara adalah rangkaian tindakan yang direncanakan dengan tatanan, aturan, tanda, atau simbol kebesaran tertentu. Pelaksanaan upacara menggunakan cara-cara yang ekspresif dari hubungan sosial terkait dengan suatu tujuan atau peristiwa yang penting. Upacara umumnya dibedakan menjadi upacara kenegaraan, upacara adat dan upacara keagamaan.[1] Peringatan kehidupan Upacara dapat menandai penyambutan dalam pekerjaan atau kehidupan seseorang, seper...

此生者传记条目需要补充更多可供查證的来源。 (2018年7月17日)请协助補充可靠来源,无法查证的在世人物内容将被立即移除。 井柏然男艺人罗马拼音Jing Boran昵称井寶、小井、寶兒、然然、小井先生国籍 中华人民共和国民族汉族籍贯遼寧瀋陽出生 (1989-04-19) 1989年4月19日(35歲) 中国遼寧省瀋陽市职业演員、歌手、模特兒语言普通话、东北话、英语音乐类型華語流行音...

Geography of SpainContinentEuropeRegionSouthern EuropeNorth AfricaIberian PeninsulaMacaronesiaBorders1,917.8 km (1,191.7 mi)Portugal:1,214 km (754 mi)France:623 km (387 mi)Andorra:63.7 km (39.6 mi)Morocco (Melilla): 9.6 km (5.97 mi) Morocco (Ceuta): 6.3 km (3.91 mi) Gibraltar: 1.2 km (0.75 mi)Highest pointTeide (Canary Islands) 3,718 m (12,198 ft) Mulhacén (Iberian Peninsula) 3,477 m (11,407 ft)Lowest ...

Bond issued by a government Government investment redirects here. For investment by governments, see Government spending. This article needs additional citations for verification. Please help improve this article by adding citations to reliable sources. Unsourced material may be challenged and removed.Find sources: Government bond – news · newspapers · books · scholar · JSTOR (July 2008) (Learn how and when to remove this message) U.S. government bond:...

The LessonTitolo originaleThe Lesson Lingua originaleinglese Paese di produzioneStati Uniti d'America Anno1913 Durata300 metri (1 rullo) Dati tecniciB/Nrapporto: 1,33 : 1film muto Generedrammatico RegiaOscar Eagle SceneggiaturaJ. Edward Hungerford ProduttoreWilliam Nicholas Selig Casa di produzioneSelig Polyscope Company Interpreti e personaggi Winifred Greenwood: Rosie Lee Jack Nelson: Jerry Brooks, il fidanzato di Rosie Frank Weed: Daddy Lee, il padre di Rosie Timmy Sheehan: il fratell...


